北海道大学大学院先端生命科学研究院の比能 洋教授と同大学大学院生命科学院博士後期課程の浦上彰吾氏の研究グループは、グラム陰性菌の表面を覆うO抗原多糖の繰り返し構造を迅速に同定することが可能な技術を開発しました。
腸管出血性大腸菌における「O157」などの記号は、種内の多様性を識別する血清型を表し、食中毒や感染症の経路判定などに広く使用されています。しかし、その候補血清型が多すぎるため、従来の陽性・陰性判定技術ではO抗原「糖鎖型」情報までを迅速に判定することが困難でした。
研究グループは、同グループが開発した糖鎖選択的イオン化によるMALDIグリコタイピング技術を活用し、マトリックス支援レーザー脱離イオン化飛行時間型質量分析法(MALDI-TOF MS)によるグラム陰性菌O抗原多糖構造の迅速同定法を開発しました。この技術により、同一O抗原血清型内の亜型構造の同定、未知のO抗原「糖鎖型」を直接発見、大腸菌O157のような「種」と「糖鎖型」の並列迅速判定が可能であることを実証しました。
この成果は、微生物種に加え、O抗原等の糖鎖型まで迅速同定可能な技術基盤を提供します。すでにMALDI-TOF MS技術の普及が進んでいる臨床微生物学分野にとどまらず、食品産業、物流の安全保障など、幅広い分野での活用が期待されます。
本研究成果は、2024年6月3日(月)公開のScientific Reports誌にオンライン掲載されました。
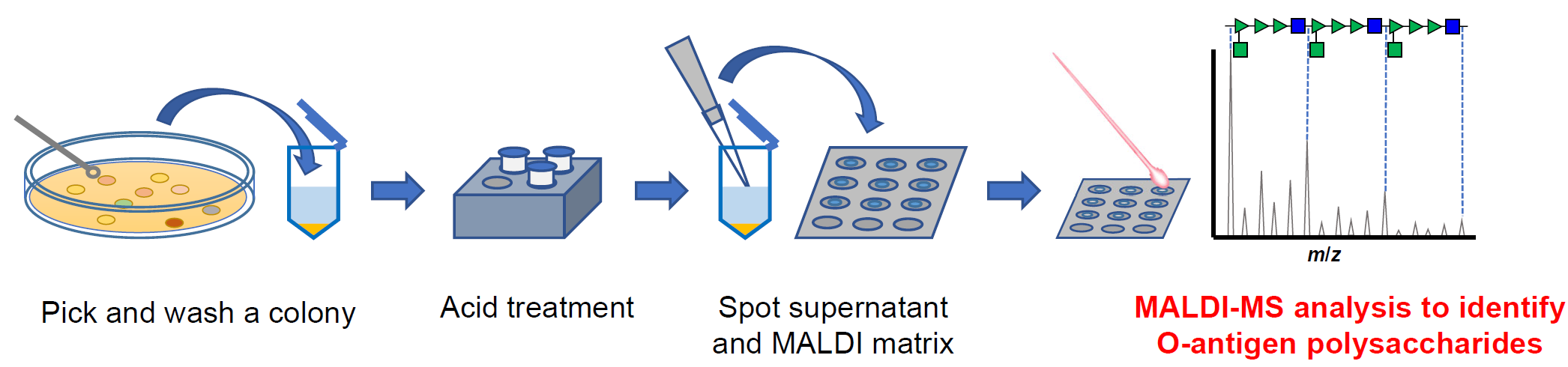
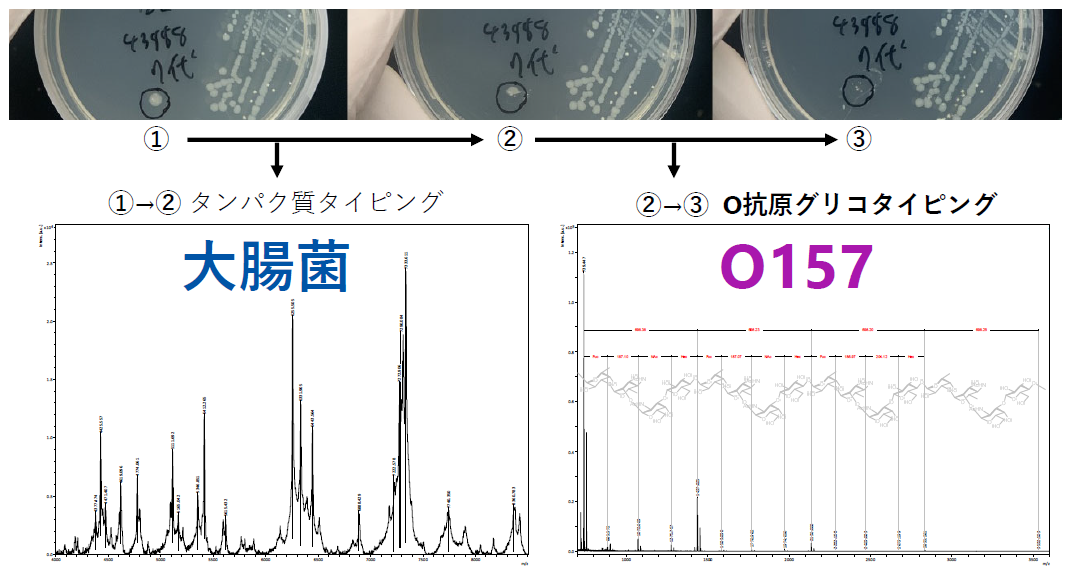
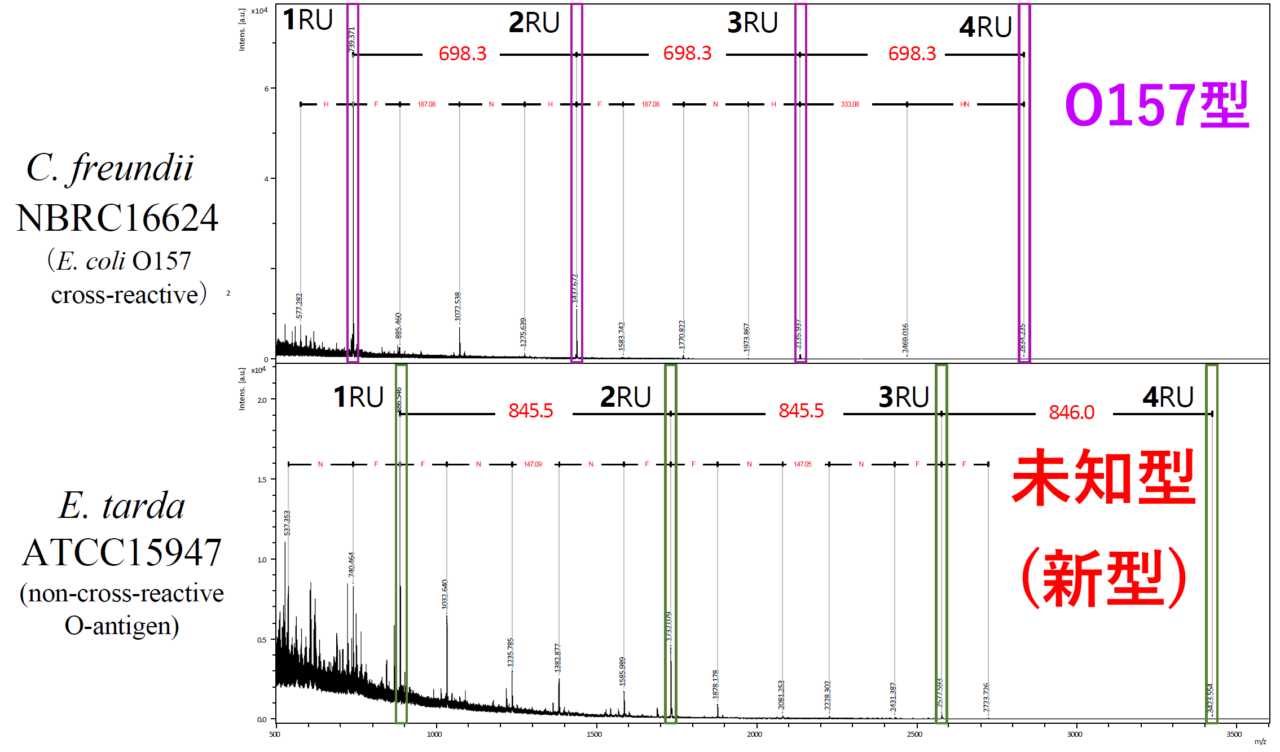
論文名 MALDI glycotyping of O-antigens from a single colony of gram-negative bacteria
(グラム陰性菌単コロニーからのO抗原MALDIグリコタイピング)
著者名 浦上彰吾1、比能 洋1,2(1北海道大学大学院生命科学院、2北海道大学大学院先端生命科学研究院)
雑誌名 Scientific Reports
DOI 10.1038/s41598-024-62729-1
公表日 2024年6月3日(月)(オンライン公開)